Entrada de Datos
1.8. Entrada de Datos al Sistema
R es muy versátil en cuanto a las fuentes de datos que pueden traerse al ambiente de computación.
Aparte de la entrada manual de datos, por ejemplo utilizando vectores para crear luego matrices o data frames, hay procedimientos para importar datos de diferentes fuentes y sus formatos.
RStudio provee el menú Import Dataset para diversos tipos de archivos desde los cuales importar datos al sistema. Más adelante utilizaremos uno de ellos.
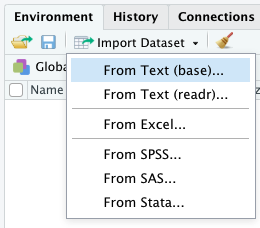
Menú Import Dataset
1.8.1. Entrada de datos manuales con un editor del sistema
Al finalizar esta sección podrás entrar y editar datos usando un editor del sistema.
Una manera de realizar la entrada manual de datos en R, es usando un editor mediante la función edit. Esta función trabaja en R solamente, por lo cual debemos usarla en la Console.
# creamos la estructura del data frame sin datos
misdatos <- data.frame(id = character(0), altura = numeric(0))
# para iniciar la entrada de datos manuales podemos escribir en la consola:
# misdatos <- edit(misdatos)
# al terminar se hace clic en Quit
# revisamos si tenemos los datos
misdatos## [1] id altura
## <0 rows> (or 0-length row.names)# si queremos continuar entrando datos o editando usamos:
# misdatos <- edit(misdatos) en la consolaEste método es fácil y rápido cuando se tienen pocos datos y no están en un documento digital. Cuando los datos son muchos, y se encuentran en un documento digital, es conveniente usar los procedimientos que se describen a continuación, para algunos formatos digitales.
1.8.2. Datos contenidos en paquetes.
Al completar esta sección podrás traer al sistema datos que se encuentran encapsulados en paquetes.
Algunos paquetes pueden contener datos para ser utilizados como ejemplos para conocer las funcionalidades del paquete o simplemente son paquetes de datos reusables. A continuación utilizaremos el paquete healthcareai para mostrar como obtener los datos contenidos en el mismo. Los datos a utilizar corresponden a un estudio de salud, realizado en 768 mujeres de más de 21 años, del grupo indígena Pima ( Akimel O’odham) del sur de Arizona y noroeste de México.
# activar el paquete healthcareai previamente instalado
library(healthcareai)
# obtener la lista de datos que contiene el paquete
data(package = "healthcareai")Data sets in package ‘healthcareai’:
pima_diabetes Patient diabetes dataset
pima_meds Patient medications dataset
Una vez activado el paquete con library, los datos del mismo están disponibles, y podemos visualizarlo y usarlos para análisis.
# la función head muestra las primeras 6 filas de datos
head(pima_diabetes)## # A tibble: 6 x 10
## patient_id pregnancies plasma_glucose diastolic_bp skinfold insulin
## <int> <int> <int> <int> <int> <int>
## 1 1 6 148 72 35 NA
## 2 2 1 85 66 29 NA
## 3 3 8 183 64 NA NA
## 4 4 1 89 66 23 94
## 5 5 0 137 40 35 168
## 6 6 5 116 74 NA NA
## # … with 4 more variables: weight_class <chr>, pedigree <dbl>, age <int>,
## # diabetes <chr>
1.8.3. Datos en archivos del sistema (.Rdata)
Al finalizar esta sección podrás guardar y cargar al sistema el contenido de un archivo .Rdata
R provee una manera muy práctica de guardar varios objetos del sistema en un solo archivo usando la función save. Este tipo de archivo puede luego ser cargado al sistema en un solo paso usando la función load. A continuación vamos a crear un archivo .Rdata y luego cargarlo al sistema.
# vector vc
vc <- c(1:20)
# matriz mx
mx <- matrix(1:9, nrow = 3, byrow = TRUE)
# resultado de función trigonométrica
ft <- sin(pi/2)
# guardar como .Rdata
save(mx, vc, ft, file = "data/archivo.Rdata")
# borramos los objetos guardados en la memoria del sistema
rm(mx,vc,ft)
# cargamos el archivo creado y vemos su contenido
print(load("data/archivo.Rdata"))## [1] "mx" "vc" "ft"
1.8.4. Importar datos desde un archivo en formato de texto delimitado.
Al completar esta sección podrás importar datos de documentos en formato .csv y similares
Una manera de guardar datos, que puedan ser utilizados por diferentes programas o lenguajes, es mediante archivos de texto delimitado. Este tipo de archivo utiliza menos memoria y no tienen incorporadas peculiaridades de formato, específicas de los programas que los generan.
Para importar datos de estos archivos podemos utilizar la función genérica read.table, usando el argumento sep = “,” para indicar que se trata de datos separados por coma. El objeto resultante es un data frame. El archivo que vamos a usar en este ejemplo es: death_small_cities.csv
# usamos read.table y los parámetros header y sep
mort_ciudades <- read.table("data/death_small_cities.csv",
header = TRUE,
sep = ",")
# para ver las primeras 10 filas de datos en el archivo
head(mort_ciudades, 10)## death1K doctor100K hosp100K income1K density
## 1 8.0 78 284 9.1 109
## 2 9.3 68 433 8.7 144
## 3 7.5 70 739 7.2 113
## 4 8.9 96 1792 8.9 97
## 5 10.2 74 477 8.3 206
## 6 8.3 111 362 10.9 124
## 7 8.8 77 671 10.0 152
## 8 8.8 168 636 9.1 162
## 9 10.7 82 329 8.7 150
## 10 11.7 89 634 7.6 134
Para separar datos delimitados por tabuladores ( tabs), usamos sep = ". Si el archivo no contiene nombres de las variables en la primera línea, se pueden agregar usando el argumento col.names = vector-con-los-nombres.
La función read.csv también se usa para importar datos delimitados, y está basada en read.table, pero es exclusiva para datos separados por coma (archivos que tienen la extensión .csv).
mort_ciudades2 <- read.csv("data/death_small_cities.csv")
# para ver las primeras 10 líneas de datos
head(mort_ciudades2, 10)## death1K doctor100K hosp100K income1K density
## 1 8.0 78 284 9.1 109
## 2 9.3 68 433 8.7 144
## 3 7.5 70 739 7.2 113
## 4 8.9 96 1792 8.9 97
## 5 10.2 74 477 8.3 206
## 6 8.3 111 362 10.9 124
## 7 8.8 77 671 10.0 152
## 8 8.8 168 636 9.1 162
## 9 10.7 82 329 8.7 150
## 10 11.7 89 634 7.6 134
Copiar datos desde una tabla
Al estilo Copy & Paste
La función read.table permite pegar ( paste) la información que se encuentra en el clipboard luego de copiar ( copy) desde una tabla u hoja de cálculo, Excel por ejemplo.
pegar <- read.table(file = "clipboard",
sep = "\t",
header = TRUE)
1.8.5. Importar desde hojas de cálculos.
Al finalizar esta sección podrás importar datos de hojas de cálculo, como Excel, usando menú y códigos R.
Para importar datos de algún programa de hoja de cálculo (Excel, OpenOffice, Google Sheets) la manera estándar consiste en exportar los datos a un archivo en formato .csv (comma separated values), y luego importarlo a R usando read.csv. Existen paquetes que proveen funciones para importar datos desde archivos Excel, uno de ellos, y asociado a RStudio es readxl; se puede usar en código o mediante el menú Import Dataset.
Paquete readxl y función read_excel(..)
Combinación de menú y código para importar datos desde Excel
Antes de iniciar el proceso de importar debe estar instalado el paquete readxl, siguiendo el procedimiento que se describió anteriormente. En una página R Script o en la consola, activamos el paquete usando library(readxl).
Debemos asegurar que el archivo que vamos a importar se encuentre en el directorio de trabajo o uno conocido. A continuación un enlace para descargar el archivo del ejemplo de esta sección:
Ahora utilizaremos el menú Import Dataset en la ventana Environment:
Hacer clic en From Excel… y se abrirá la ventana de importación.
Lo primero es localizar el archivo a importar, usando Browse…, y si el archivo se encuentra en el directorio de trabajo, este debe aparecer en la lista de archivos disponibles, o dentro de una de las carpetas de este directorio.
Una vez seleccionado el archivo, tus datos deben aparecer dentro de la ventana de importación. Si no aparecen los datos que esperas, verifica en Sheet: si tienes seleccionada la hoja de datos que deseas.
Verifica si aparecen los nombres de las columnas, y si los datos son de la clase que se esperan ( numeric y double, o character). Si los nombres de la columnas no aparecen como se esperan, verificar que First Row as Names esté marcado.
Puedes cambiar el nombre del data.frame que vas a crear, usando Name:.
Quita la marca de Open Data Viewer, y copia el código que se encuentra en la ventana de Code Preview:, y pégalo en tu R Script.
Corre el código copiado, y usando head(nombre_de_data.frame) debes poder ver las seis primeras filas de datos de tu data frame.
VIDEO
# activación del paquete
library(readxl)
# código de carga de datos del archivo
mort_ciudades3 <- read_excel("data/death_small_cities.xlsx",
sheet = "data")
head(mort_ciudades3, 10)## # A tibble: 10 x 5
## death1K doctor100K hosp100K income1K density
## <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 8 78 284 9.10 109
## 2 9.30 68 433 8.70 144
## 3 7.5 70 739 7.20 113
## 4 8.90 96 1792 8.90 97
## 5 10.2 74 477 8.30 206
## 6 8.30 111 362 10.9 124
## 7 8.80 77 671 10 152
## 8 8.80 168 636 9.10 162
## 9 10.7 82 329 8.70 150
## 10 11.7 89 634 7.60 1341.8.6. Importar datos de la web
Al finalizar esta sección podrás importar datos de sitios en la web que provean acceso a archivos de datos
Algunos sitios públicos de la web permiten descargar algunos de sus datos. Podemos utilizar el enlace ( URL) para descargar los datos directamente a nuestro ambiente de trabajo. Usaremos como ejemplo, datos disponibles en la página del CDC - National Center for Health Statistics (https://data.cdc.gov/NCHS/), y específicamente los datos provisionales de muertes asociadas a COVID-19, por edad, sexo y estado o territorio. El paquete rio provee la función download.file para realizar la descarga de un archivo en formato .csv, que luego podemos cargar al sistema con read.csv
# activar rio
library(rio)
# copiar la dirección del archivo obtenidad de la página web
covid <- "https://data.cdc.gov/api/views/9bhg-hcku/rows.csv?accessType=DOWNLOAD"
# descargar los datos con download.file
download.file(covid, "data/coviddata.csv")
# importar los datos al sistema usando read.csv
coviddeath <- read.csv("data/coviddata.csv")
# primeras 6 filas de los datos
head(coviddeath)## Data.As.Of Start.Date End.Date Group Year Month State Sex
## 1 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## 2 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## 3 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## 4 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## 5 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## 6 06/16/2021 01/01/2020 06/12/2021 By Total NA NA United States All Sexes
## Age.Group COVID.19.Deaths Total.Deaths Pneumonia.Deaths
## 1 All Ages 590091 4791858 520776
## 2 Under 1 year 81 26746 288
## 3 0-17 years 322 46977 773
## 4 1-4 years 37 4859 156
## 5 5-14 years 112 7811 222
## 6 15-24 years 962 50406 1084
## Pneumonia.and.COVID.19.Deaths Influenza.Deaths
## 1 289584 9161
## 2 12 21
## 3 61 183
## 4 6 64
## 5 24 77
## 6 381 81
## Pneumonia..Influenza..or.COVID.19.Deaths Footnote
## 1 829164
## 2 378
## 3 1217
## 4 251
## 5 387
## 6 1741